3 de septiembre de 2021
Descubren la molécula que permite al coronavirus entrar a las células
Los científicos han buscado desentrañar los secretos de los mecanismos que permiten al coronavirus ingrese e infecte las células humanas sanas.
Una colaboración entre investigadores de universidades de EEUU y el uso de supercomputadoras permitió encontrar la puerta que hace que el virus ingrese en el organismo. Cómo se aplicará el hallazgo
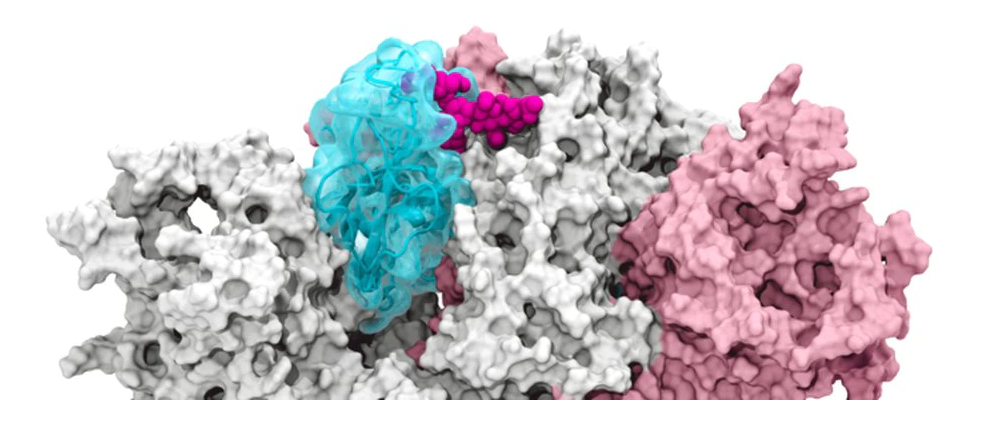
Desde los primeros días de la pandemia de COVID-19, los científicos han buscado desentrañar los secretos de los mecanismos que permiten al coronavirus ingrese e infecte las células humanas sanas. Un equipo de investigadores de universidades de Estados Unidos descubrió el mecanismo por el cual unas moléculas que forman un residuo azucarado alrededor de los bordes de la proteína Espiga del coronavirus- actúan como puertas de entrada a la infección.
La proteína “Espiga” o “espícula” (“Spike” en inglés) del coronavirus ya entró en el vocabulario popular tras más de un año y medio de pandemia. El año pasado, a principios de la pandemia, Rommie Amaro, la investigadora en química biofísica computacional de la Universidad de California en San Diego, fue clave para aportar una visualización detallada de la proteína Espiga del coronavirus SARS-CoV-2 que se adhiere eficazmente a los receptores de las células humanas.
Ahora, Amaro y sus colegas de investigación de la Universidad de California en San Diego, la Universidad de Pittsburgh, la Universidad de Texas en Austin, la Universidad de Columbia y la Universidad de Wisconsin-Milwaukee, de los Estados Unidos han revelado cómo los glicanos, que son las moléculas situadas alrededor de los bordes de la proteína Espiga- actúan como puertas de entrada a la infección.
La investigación fue publicada el 19 de agosto en la revista Nature Chemistry. La doctora Amaro es coautora del trabajo junto con Lillian Chong de la Universidad de Pittsburgh. La primera autora es una estudiante de posgrado de la Universidad de California en San Diego, Terra Sztain. También participó la becaria posdoctoral Surl-Hee Ahn.
“Esencialmente, hemos descubierto cómo se abre realmente la Espiga y se infecta”, dijo Amaro. “Hemos revelado un importante secreto de la proteína espiga en su forma de infectar las células. Sin esta puerta, el virus queda básicamente incapacitado para la infección”.
La investigadora sostuvo que el descubrimiento de la puerta por parte del equipo de investigación abre posibles vías de nuevas terapias para contrarrestar la infección por el coronavirus. Si las puertas de las moléculas glicanos pudieran bloquearse farmacológicamente en la posición cerrada, se impediría efectivamente que el virus se abriera para entrar e infectarse.
El recubrimiento de glicanos de la proteína Espiga ayuda a engañar al sistema inmune humano, ya que se presenta como nada más que un residuo azucarado. Las tecnologías anteriores que obtenían imágenes de estas estructuras mostraban los glicanos en posiciones estáticas abiertas o cerradas, lo que inicialmente no despertó mucho interés entre los científicos.
Pero las simulaciones de supercomputación permitieron a los investigadores estadounidenses desarrollar películas dinámicas que revelaban las puertas de los glicanos activándose de una posición a otra. De esta manera, se aportó una pieza sin precedentes de la historia de la infección.
“Pudimos ver realmente la apertura y el cierre”, dijo Amaro. “Esa es una de las cosas más interesantes que ofrecen estas simulaciones: la posibilidad de ver películas muy detalladas. Cuando las ves, te das cuenta de que estás viendo algo que de otro modo habríamos ignorado. Si miras sólo la estructura cerrada, y luego miras la estructura abierta, no parece nada especial. Sólo porque capturamos la película de todo el proceso se ve realmente cómo funciona”, agregó.
“Las técnicas estándar habrían necesitado años para simular este proceso de apertura, pero con las herramientas de simulación avanzada de mi laboratorio, el “conjunto ponderado”, pudimos capturar el proceso en sólo 45 días”, dijo la doctora Chong.
Las simulaciones de alta intensidad computacional se ejecutaron primero en Comet, en el Centro de Supercomputación de San Diego, en la UC San Diego, y después en Longhorn, en el Centro de Computación Avanzada de Texas, en la UT Austin. Esta potencia de cálculo proporcionó a los investigadores vistas a nivel atómico del dominio de unión al receptor de la proteína de Espiga, o RBD, desde más de 300 perspectivas.
Las investigaciones revelaron que el glicano “N343” es el eje que hace que el receptor RBD de la proteína Espiga pase desde la posición “abajo” a la de “arriba” para permitir el acceso al receptor ACE2 de la célula huésped. Los investigadores describen la activación del glicano N343 como un mecanismo similar a una “palanca molecular”.
Además, Jason McLellan, profesor asociado de biociencias moleculares en la UT Austin, y su equipo crearon variantes de la proteína de la Espiga y probaron para ver cómo la falta de la puerta del glicano afectaba a la capacidad de apertura del receptor RBD. “Demostramos que sin esta puerta, el recepto RBD de la proteína Espiga no puede adoptar la conformación que necesita para infectar las células”, aclaró McLellan.
En el trabajo publicado en Nature Chemistry, los autores estudian mediante simulaciones el proceso por el cual que la proteína Espiga expone la porción de su estructura que interacciona con el receptor presente en las células del huésped, comentó a Infobae Karina Mariño, investigadora del Instituto de Biología y Medicina Experimental del Conicet. “Los autores encuentran que un glicano, llamado N343, es clave para activar ese proceso, y por lo tanto, esencial para la interacción con el receptor y entrada del virus. Los resultados presentados nos ayudan a comprender mejor el proceso de entrada a la célula del coronavirus, y en particular, la importancia de la glicosilación de la proteína Espiga. Este conocimiento, si bien aún en el área de investigación básica, es clave para seguir buscando maneras de impedir la invasión viral”.
Mariano Pérez Filgueira, investigador en virología del Conicet, y miembro de la Sociedad Argentina de Virología, comentó el hallazgo en los Estados Unidos. “La investigación publicada en Nature Chemistry aportó un blanco potencial para una potencial terapia: se podría interferir de alguna forma con el proceso de entrada del coronavirus en las células humanas”.
Para el investigador argentino, el descubrimiento podría ser aplicado al desarrollo de tratamientos con fármacos antivirales. “Los antivirales pueden actuar en diferentes etapas del mecanismo de entrada, replicacion y salida del virus. Con el nuevo conocimiento, se podría apuntar al desarrollo de un antiviral que interfiera en el paso del contacto de la proteína Spiga con el receptor celular”, afirmó Pérez Filgueira.
En junio pasado, el gobierno de los Estados Unidos había anunciado la inversión de 3.200 millones de dólares en el desarrollo de píldoras antivirales contra la enfermedad COVID-19. Anthony Fauci, director del Instituto Nacional de Alergias y Enfermedades Infecciosas y uno de los principales promotores del programa, dijo en junio que esperaba que los pacientes con coronavirus puedan ir a buscar sus pastillas antivirales a una farmacia apenas sepan que son positivos en el PCR o cuando comiencen a desarrollar síntomas compatibles con COVID-19.
“Me despierto por la mañana, no me encuentro muy bien, se me va el sentido del olfato y del gusto, me duele la garganta. Llamo a mi médico y le digo: ‘Tengo COVID y necesito una receta’”, detalló el doctor Fauci sobre cómo podría ser el uso de antivirales efectivos en el futuro contra el COVID-19.
COMPARTIR:
Notas Relacionadas
Comentarios
Aun no hay comentarios, sé el primero en escribir uno!






























